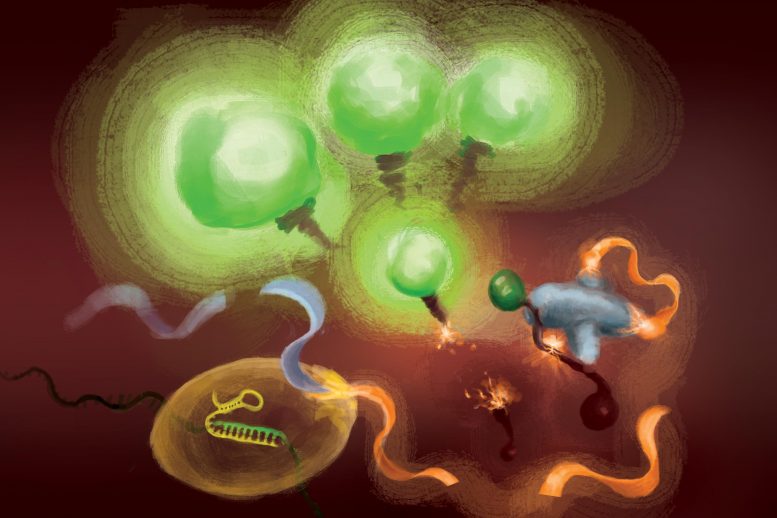
Las reacciones involucradas en la prueba FIND-IT para detectar la infección por el virus del SARS-CoV-2. Cuando la enzima Cas13 (izquierda) se une a su ARN objetivo, corta una molécula (naranja y gris) para liberar un activador (naranja) que sobrecarga la nucleasa Csm6 (centro inferior) para escindir y liberar marcadores fluorescentes que se iluminan (verde). y señalar la presencia de ARN viral. Crédito: Cortesía de Margaret L. Liu, Facultad de Medicina Pritzker de la Universidad de Chicago
Pruebas rápidas y frecuentes para COVID-19 es fundamental controlar la propagación de los brotes, especialmente cuando surgen nuevas variantes más transmisibles.
Si bien la prueba de diagnóstico COVID-19 estándar de oro de hoy, que utiliza qRT-PCR, reacción cuantitativa en cadena de la polimerasa con transcriptasa inversa (PCR), es extremadamente sensible y detecta hasta una copia de ARN por microlitro, requiere de equipos especializados, una autonomía de varias horas y un laboratorio centralizado. Como resultado, la prueba generalmente toma al menos uno o dos días.
Un equipo de investigación dirigido por científicos de los laboratorios de Jennifer Doudna, David Savage y Patrick Hsu en el Universidad de California, Berkeley, tiene como objetivo desarrollar una prueba de diagnóstico que sea mucho más rápida y fácil de implementar que la qRT-PCR. Ahora ha combinado dos tipos diferentes de enzimas CRISPR para crear una prueba que puede detectar pequeñas cantidades de ARN viral en menos de una hora. Doudna compartió el Premio Nobel de Química 2020 por la invención de la edición del genoma CRISPR-Cas9.
Aunque la nueva técnica aún no está en la etapa en la que compite con la sensibilidad de la qRT-PCR, que puede detectar solo unas pocas copias del virus por microlitro de líquido, ya es capaz de detectar niveles de ARN viral, alrededor de 30 copias por microlitro. – suficiente para controlar la población y limitar la propagación de infecciones.
«No se requiere sensibilidad de PCR para detectar y diagnosticar sustancialmente COVID-19 en la comunidad si la prueba es lo suficientemente asequible y lo suficientemente rápida», dijo el coautor David Savage, profesor de biología celular y molecular. «Nuestra esperanza era llevar la bioquímica lo más lejos posible hasta el punto en que pudiera imaginarse un formato muy conveniente en un entorno donde las pruebas son posibles todos los días, por ejemplo, a la entrada del trabajo».
Los investigadores informaron sus hallazgos el 5 de agosto de 2021 en la revista. Biología de la naturaleza química.

Tina Liu y Jennifer Doudna fuera del edificio IGI el día en que Doudna ganó el Premio Nobel de Química 2020.Crédito: UC Berkeley foto por Brittany Hosea-Small
Varias pruebas basadas en CRISPR han sido aprobadas para uso de emergencia por la Administración de Alimentos y Medicamentos, pero todas requieren una etapa inicial en la que el ARN viral se amplifica para que la señal de detección, que implica la liberación de una molécula fluorescente que brilla bajo luz azul, es lo suficientemente brillante para ver. Aunque esta amplificación inicial aumenta la sensibilidad del ensayo a un nivel similar al de la qRT-PCR, también introduce pasos que hacen que el ensayo sea más difícil de realizar fuera de un laboratorio.
El equipo dirigido por UC Berkeley buscó lograr una sensibilidad y velocidad útiles sin sacrificar la simplicidad de la prueba.
«Para las aplicaciones de punto de atención, se desea tener una respuesta rápida para que las personas puedan saber rápidamente si están infectadas o no, por ejemplo, antes de abordar un vuelo o visitar a familiares», dijo la líder del equipo Tina Liu, investigadora en El laboratorio de Doudna en el Innovative Genomics Institute (IGI), un centro centrado en CRISPR que involucra a científicos de UC Berkeley y UC San Francisco.
Además de tener un paso adicional, otra desventaja de la amplificación inicial es que debido a que produce miles de millones de copias de ARN viral, existe una mayor probabilidad de contaminación cruzada entre muestras de pacientes. La nueva técnica desarrollada por el equipo revierte esta situación y, en cambio, aumenta la señal fluorescente, eliminando una fuente importante de contaminación cruzada.
La técnica de no amplificación, a la que llaman Detección rápida integrada de nucleasas en tándem (FIND-IT), podría permitir pruebas de diagnóstico rápidas y económicas para muchas otras enfermedades infecciosas.
«Si bien comenzamos este proyecto con el propósito expreso de impactar COVID-19, creo que esta técnica en particular podría ser aplicable a más que esta pandemia porque, en última instancia, CRISPR es programable», dijo Liu. “Entonces, podría cargar la enzima CRISPR con una secuencia dirigida al virus de la influenza o VIH o cualquier tipo de virus de ARN y el sistema tiene el potencial de funcionar de la misma manera. Este documento realmente establece que esta bioquímica es una forma más fácil de detectar ARN y tiene la capacidad de detectar ese ARN en un marco de tiempo sensible y rápido que puede estar abierto a futuras aplicaciones en el diagnóstico en el punto de atención «.
Actualmente, los investigadores están construyendo un diagnóstico de este tipo utilizando FIND-IT, que incluiría pasos para recolectar y procesar muestras y realizar la prueba en un dispositivo microfluídico compacto.
Uso de proteínas Cas en tándem
Para eliminar la amplificación del objetivo de la ecuación, el equipo utilizó una enzima CRISPR, Cas13, para detectar primero el ARN viral y otro tipo de proteína Cas, llamada Csm6, para amplificar la señal de fluorescencia.
Cas13 es una tijera multiusos para cortar ARN; una vez que se une a su secuencia objetivo, especificada por un ARN guía, está listo para escindir una amplia gama de otras moléculas de ARN. Esta actividad de escisión activada por la diana se puede aprovechar para acoplar la detección de una secuencia de ARN específica a la liberación de una molécula indicadora fluorescente. Sin embargo, por sí solo, Cas13 puede tardar horas en generar una señal detectable cuando están presentes cantidades muy bajas de ARN objetivo.
La intuición de Liu fue usar Csm6 para amplificar el efecto de Cas13. Csm6 es una enzima CRISPR que detecta la presencia de pequeños anillos de ARN y se activa para escindir una amplia gama de moléculas de ARN en las células.
Para mejorar la detección de Cas13, ella y sus colegas diseñaron una molécula activadora especialmente diseñada que se corta cuando Cas13 detecta ARN viral. Un fragmento de esta molécula puede unirse y activar Csm6 para cortar y liberar una molécula fluorescente brillante de una pieza de ARN. Normalmente, la molécula activadora es rápidamente degradada por Csm6, lo que limita la cantidad de señal fluorescente que puede generar. Liu y sus colegas idearon una forma de modificar químicamente el activador para que esté protegido de la degradación y pueda sobrecargar Csm6 para cortar y liberar repetidamente moléculas fluorescentes unidas al ARN. Esto da como resultado una sensibilidad 100 veces mejor que el activador original.
«Cuando se activa Cas13, escinde este pequeño activador, eliminando un segmento que lo protege», dijo Liu. “Ahora que se libera, puede activar muchas moléculas diferentes de esa segunda enzima, Csm6. Y así, un objetivo reconocido por Cas13 no solo conduce a la activación de la capacidad de uno para cortar el ARN; conduce a la generación de muchas más enzimas activas que luego pueden degradar aún más reporteros fluorescentes ”.
El equipo de investigadores también incorporó una combinación optimizada de ARN guía que permite un reconocimiento más sensible del ARN viral por Cas13. Cuando esto se combinó con Csm6 y su activador, el equipo pudo detectar hasta 31 copias por microlitro de SARS-CoV-2 ARN en solo 20 minutos.
Los investigadores también agregaron ARN extraído de muestras de pacientes a la prueba FIND-IT en un cartucho de microfluidos, para ver si esta prueba podría adaptarse para ejecutarse en un dispositivo portátil. Usando un pequeño dispositivo con una cámara, pudieron detectar el ARN del SARS-CoV-2 extraído de muestras de pacientes con una sensibilidad que capturaría las infecciones por COVID-19 en su punto máximo.
«Este enfoque de nucleasa en tándem, Cas13 más Csm6, combina todo en una sola reacción a una sola temperatura, 37 grados Centígrado, por lo que no requiere calentamiento a alta temperatura o múltiples pasos, como se requiere para otras técnicas de diagnóstico «, dijo Liu.» Creo que esto abre oportunidades para pruebas más rápidas y simples que pueden lograr una sensibilidad comparable a otras técnicas actuales y potencialmente podrían lograr sensibilidades aún mayores en el futuro «.
El desarrollo de este método sin amplificación para la detección de ARN es el resultado de una reorientación de la investigación dentro del IGI a medida que comenzó la pandemia hacia los problemas de diagnóstico y tratamiento de COVID-19. Finalmente, cinco laboratorios de UC Berkeley y dos laboratorios de UCSF participaron en este proyecto de investigación, uno de los muchos dentro del IGI.
“Cuando comenzamos, esperábamos crear algo que logre la paridad con la PCR, pero que no requiera amplificación, ese iba a ser el sueño”, dijo Savage, quien fue el investigador principal del proyecto. “Y desde el punto de vista de la sensibilidad, teníamos una brecha de unas diez mil veces para saltar. Lo hemos hecho unas mil veces; lo bajamos en unos tres órdenes de magnitud. Entonces, casi estamos allí. En abril pasado, cuando estábamos empezando a trazar un mapa, parecía casi imposible «.
Referencia: «Detección acelerada de ARN usando nucleasas CRISPR en tándem» por Tina Y. Liu, Gavin J. Knott, Dylan CJ Smock, John J. Desmarais, Sungmin Son, Abdul Bhuiya, Shrutee Jakhanwal, Noam Prywes, Shreeya Agrawal, María Díaz de León Derby, Neil A. Switz, Maxim Armstrong, Andrew R. Harris, Emeric J. Charles, Brittney W. Thornton, Parinaz Fozouni, Jeffrey Shu, Stephanie I. Stephens, G. Renuka Kumar, Chunyu Zhao, Amanda Mok, Anthony T. Iavarone, Arturo M. Escajeda, Roger McIntosh, Shineui Kim, Eli J. Dugan, IGI Testing Consortium, Katherine S. Pollard, Ming X. Tan, Melanie Ott, Daniel A. Fletcher, Liana F. Lareau, Patrick D. Hsu, David F. Savage y Jennifer A. Doudna, 5 de agosto de 2021, Biología de la naturaleza química.
DOI: 10.1038 / s41589-021-00842-2
El trabajo fue apoyado por la Agencia de Proyectos de Investigación Avanzada de Defensa (N66001-20-2-4033). Los coautores del artículo incluyen miembros de laboratorio de Jennifer Doudna, David Savage, Patrick Hsu, Liana Lareau y Daniel Fletcher en UC Berkeley; Gavin Knott de la Universidad de Monash en Australia; Melanie Ott y Katherine Pollard de los Institutos Gladstone y UCSF; y Ming Tan de Wainamics, una empresa de investigación y desarrollo en Pleasanton, California, que fabrica dispositivos de microfluidos. Doudna, fundador del IGI y actualmente presidente y presidente de la junta directiva del IGI, es el presidente del Canciller de Li Ka Shing en UC Berkeley y profesor de química y biología celular y molecular. Hsu, Lareau y Fletcher son profesores del Departamento de Bioingeniería.
También te puede interesar
-
Dormir bien el fin de semana puede reducir en una quinta parte el riesgo de sufrir enfermedades cardíacas: estudio | Cardiopatía
-
Una nueva investigación sobre la falla megathrust indica que el próximo gran terremoto puede ser inminente
-
Caso de Mpox reportado en la cárcel del condado de Las Vegas
-
SpaceX lanzará 21 satélites Starlink en el cohete Falcon 9 desde Cabo Cañaveral – Spaceflight Now
-
SpaceX restablece el lanzamiento pospuesto de Polaris Dawn, una misión espacial comercial récord
